La inocuidad alimentaria es un tema prioritario de salud pública. En Chile, el Ministerio de Salud establece políticas alimentarias destinadas a la protección y el fomento de la salud de los consumidores, mientras la Agencia Chilena para la Inocuidad y Calidad Alimentaria (ACHIPIA) dependiente del Ministerio de Agricultura, formula la Política Nacional de Inocuidad y Calidad Alimentaria, y coordina a los organismos públicos y privados que participan en la cadena de producción, distribución y fiscalización, hasta llegar al consumidor. Tanto la coordinación de política pública, junto a la normativa sanitaria de los alimentos han permitido que los indicadores de salud pública y los asociados a las enfermedades de transmisión por alimentos sean de los mejores de América Latina, alcanzando los estándares internacionales.
A pesar de estos esfuerzos, durante la última década el mundo ha enfrentado numerosas crisis alimentarias, provocadas por la contaminación de los alimentos. Los brotes de enfermedades transmitidas por los alimentos han generado costos económicos, sociales y comerciales asociados a la prevención y control de ellas. Algunos de los microorganismos causantes de estos brotes son bacterias resistentes a antibióticos, compuestos que interfieren con el crecimiento o causan la muerte de las bacterias, y son utilizados para combatir enfermedades. Si bien el uso de antibióticos es necesario para establecer sistemas sustentables de agricultura, ganadería y acuicultura, en la actualidad, animales de consumo humano son catalogados como un importante reservorio de bacterias resistentes a antibióticos, y la industria de producción de alimentos se señala como una de las responsables de la problemática global de resistencia antimicrobiana. Estas bacterias resistentes a antibióticos pueden esparcirse por el medio ambiente, transmitiéndose desde los animales que los portan hacia su entorno, como granjas, centros de procesamiento y distribución, sistemas de transporte y venta. Adicionalmente, los operarios, manipuladores e intermediarios también son susceptibles de contaminarse, así como la población general vía la contaminación de suelos, cursos de agua y el consumo de los productos contaminados.
En Chile, la industria salmonera ha sido señalada nacional e internacionalmente como un agente contaminante, siendo la responsable del uso de más de 460 toneladas de antibióticos (Informe sobre Uso de Antimicrobianos en la Salmonicultura Nacional - Año 2021, SERNAPESCA, Servicio Nacional de Pesca y Acuicultura). Sin embargo, el uso de antibióticos por esta industria en Chile ha sido sustantivo para el tratamiento de enfermedades bacterianas en salmónidos, y data de varias décadas. La administración de antibióticos en la industria acuícola está fuertemente regulada por SERNAPESCA, y ocurre principalmente por medio de alimentos medicados. Como efecto colateral, parte de este alimento se libera y dispersa por el ambiente marino, al igual que las heces y subproductos de la metabolización de los antibióticos (1), los que pueden permanecer en el agua y sedimentos marinos por periodos prolongados. Como resultado de ello, numerosos estudios realizados en la Región de los Lagos han aislado una gran cantidad de bacterias resistentes a antibióticos desde los sedimentos de centros de cultivo de salmones y la microbiota intestinal de los salmónidos. El uso profiláctico y terapéutico de antibióticos no solo favorece la aparición de bacterias resistentes a antibióticos, sino también de los elementos genéticos que codifican los determinantes de resistencia, los genes de resistencia a antibióticos (GRA) (2). Mediante la secuenciación al azar de los genomas de todas las bacterias presentes en una muestra ambiental (secuenciación metagenómica) ha sido posible predecir una gran cantidad y diversidad de GRA en bacterias ambientales. Ello ha hecho suponer que el medio ambiente es un reservorio de estos genes, y por tanto un indicador del potencial de resistencia de este.
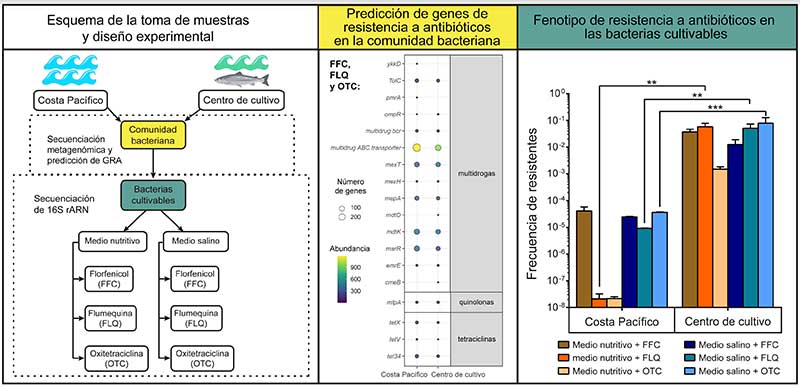
Considerando lo anterior, en nuestro laboratorio hemos adoptado una estrategia intermedia entre el cultivo de bacterias aisladas desde el ambiente y la secuenciación de ellas para identificar la presencia de GRA. Para probar nuestro protocolo, agua superficial de mar, recolectada en un sitio prístino (Costa Pacífico) y en un plantel productivo de salmónidos (Centro de cultivo), fue filtrada y la fracción bacteriana se cultivó en placas con diferentes medios de cultivos. Posteriormente, se evaluó la cantidad de bacterias resistentes obtenidas para cada sitio (ver figura panel izquierdo). Los medios estudiados fueron seleccionados por su diferencia en cantidad de nutrientes y salinidad. Los antibióticos seleccionados fueron aquellos utilizados comúnmente en salmonicultura: florfenicol (FFC), oxitetraciclina (OTC) y flumequina (FLQ). Con el fin de conocer la identidad de las bacterias ambientales cultivables, utilizamos una técnica basada en la secuenciación directa del gen 16S rARN (que actúa como una “huella dactilar” para la identificación de bacterias) del césped bacteriano (colonias bacterianas fusionadas formando una capa uniforme)) sin necesidad de aislar las colonias bacterianas.
Lo anterior nos permitió, evaluar y comparar rápidamente la selectividad de los medios de cultivo y el efecto de los diferentes antibióticos sobre la composición de las comunidades bacterianas cultivables. Sumado a ello, utilizamos secuenciación metagenómica para identificar los GRA de la comunidad bacteriana (ver leyenda de la figura) presente en cada sitio, Costa Pacífico o Centro de cultivo (ver Figura panel izquierdo). Interesantemente, la abundancia y diversidad de GRA identificados en las comunidades bacterianas fue similar para los dos sitios, sugiriendo un potencial de resistencia a antibióticos similar (ver figura panel central). Sin embargo, al evaluar la frecuencia de aparición de bacterias resistentes a antibióticos se observó una gran diferencia entre los sitios, mostrando que, a pesar de la similitud en el potencial genético de resistencia a antibióticos, la comunidad bacteriana proveniente del Centro de cultivo resultó ser fenotípicamente más resistente (ver figura panel derecho). Esta diferencia entre el fenotipo de resistencia observado, en relación al potencial genético de resistencia, ha sido señalado anteriormente como un problema de salud pública al cual debe ponerse atención. En 2016, científicos estadounidenses junto a la Asociación de Laboratorios de Salud Pública de USA hacían un llamado a continuar con el cultivo de microorganismos patógenos con el fin de mantener la vigilancia epidemiológica, indicando que el diagnóstico realizado exclusivamente mediante técnicas de secuenciación de alto rendimiento era una amenaza a la trazabilidad frente a brotes de contaminaciones alimentarias (3). Por otro lado, otros autores han señalado que una de las dificultades de la secuenciación masiva es que se observan tanto microorganismos vivos como muertos, y que ambas técnicas (secuenciación y cultivo) son complementarias a la hora de conocer la diversidad, riqueza y por, sobre todo, función de las comunidades bacterianas.
Referencias
-
Barreto F.M., da Silva M.R., Braga P.A.C., Bragotto A.P.A., Hisano H., Reyes F.G.R. Evaluation of the leaching of florfenicol from coated medicated fish feed into water. Environ. Pollut. 2018; 242:1245–1252. doi: 10.1016/j.envpol.2018.08.017.
-
Tamminen, M., Karkman, A., Lõhmus, A., Muziasari, W. I., Takasu, H., Wada, S., et al. Tetracycline resistance genes persist at aquaculture farms in the absence of selection pressure. Environ. Sci. Technol. 2010; 45:386–391. doi: 10.1021/es102725n.
-
Shea S., Kubota K.A., Maguire H., Gladbach S, Woron A., Atkinson-Dunn R., Couturier M.R., Miller M.B. Clinical microbiology laboratories' adoption of culture independent diagnostic tests is a threat to foodborne-disease surveillance in the United States. J Clin Microbiol. 2017; 55:10 –19. https:// doi.org/10.1128/JCM.01624-16.


